Gebruiker:Bas dehaan/Kladblok/Kwantitatieve polymerasekettingreactie
| Mee bezig Aan deze pagina of deze sectie wordt de komende uren of dagen nog druk gewerkt.
Klik op geschiedenis voor de laatste ontwikkelingen. |


Een kwantitatieve polymerasekettingreactie (qPCR) (Engels: quantitative Polymerase Chain Reaction), ook wel bekend als real-time PCR is een laboratoriumtechniek die wordt gebruikt in de moleculaire biologie en medische diagnostiek. Het is afgeleid van de polymerasekettingreactie (PCR). qPCR kan worden gebruikt om de amplificatie van het getargetde DNA te meten tijdens de PCR, en dus niet enkel na afloop van de kettingreactie, zoals bij een reguliere PCR het geval is.
Twee veelvoorkomende methodes voor die worden gebruikt het detecteren van het PCR-product in qPCR zijn (1) aspecifieke fluorescente kleuring dat hecht aan het dubbelstrengs DNA en (2) sequentie-specifieke DNA probes welke bestaan uit oligonucleotides die zijn gelabeld met een fluorescente reporter, welke alleen gedetecteerd kan worden nadat de probe volledig met de DNA-sequentie is gehybridiseerd.
Achtergrond[bewerken | brontekst bewerken]

Binnen alle organismen reguleren cellen de genexpressie aan de hand van de verversingssnelheid van translatie-producten (veelal Messenger-RNA). De mate van expressie van een gen in een cel kan daardoor worden bepaald aan de hand van het aantal kopieën van het RNA-transcript van het betreffende gen. Om het aantal kopiëen op een degelijke wijze te kunnen kwantificeren dient die kleine hoeveelheid RNA in een sample te worden geamplificeerd. In tegenstelling tot een DNA-sample, waarbij een polymerasekettingreactie (PCR) direct kan worden opgestart, dient in het geval van een RNA-sample het RNA eerst te worden omgezet in DNA met behulp van reverse-transcriptase.
Bij qPCR wordt voor het amplificeren de zelfde methodiek gebruikt als bij een conventionele PCR, dat wil zeggen dat er net als bij een conventionele PCR ook gebruik gemaakt wordt van tenminste een set van twee primers, deoxyribonucleotides, een geschikte bufferoplossing en thermo-stabile DNA-polymerase. Daarnaast dient een fluorofoor toegevoegd te worden aan het te amplificeren sample. Voor de PCR zelf dient een speciale thermal cycler gebruikt te worden, welke is uitgerust met sensoren om de fluorescentie op een bepaalde golflengte te kunnen meten. Dit zorgt ervoor dat de amplificatie van het product na iedere PCR cyclus gemeten kan worden. De data die hierbij gegenereerd wordt kan vervolgens worden geanalyseerd met behulp van computer software, om zo een relative gen expressie (of mRNA copy number) te kunnen bereken over verschillende samples. qPCR kan ook worden gebruikt om een DNA-sequentie te detecteren en eventuele repeats te kwantificeren.[1]
Kwantitatieve PCR wordt, net als DNA-microarrays, gezien als een moderne methode voor het bestuderen van genexpressie. Zij wordt echter beperkt door benaderingsfouten als gevolg van variatie in onder andere DNA integriteit en enzymefficiëntie tussen cellen en samples. Daarom zijn er verschillende standaardisering methodes ontwikkeld om hiervoor te compenseren. Meestal wordt daarom de genexpressie uitgedrukt in een relative verhouding tot een huishoudgen, aangezien dit genen zijn die relatief eenvoudig zijn en in een constante mate tot expressie komen, aangezien het veelal genen zijn die cruciaal zijn voor het overleven van de cel.[2][3] Deze relaltieve genexpressie stelt onderzoekers instaat om de mate van genexpressie tussen bijvoorbeeld verschillende genen of condities met elkaar te kunnen vergelijken.
Basis principes[bewerken | brontekst bewerken]
qPCR wordt uitgevoerd met behulp van een thermal cycler welke is uitgerust met de functie om ieder sample te belichten met licht van een specifieke golflengte en vervolgens de uitgestraalde fluorescentie van de fluorofoor te kunnen meten. Daarnaast dient de thermal cycler instaat te zijn om de samples snel te kunnen verhitten en verkoelen, om zo gebruik te kunnen maken van de fysisch-chemische eigenschappen van de nucleïnezuren en DNA-polymerase.
Het PCR proces bestaat over het algemeen uit een serie temperatuurwijzigingen die 25 tot 50 keer worden herhaald. Deze cycli bestaan veelal uit 3 fases: eerst de denaturatie-stap, waarin de temperatuur wordt verhoogd naar ongeveer 95°C om de dubbele helix van het DNA te verbreken; als tweede een annealing-stap, waarin, bij een temperatuur van 45°C to 65°C, de toegevoegde primer aan het DNA kan binden;[4] als derde wordt, bij een temperatuur rond de 72°C, een elongatie-stap uitgevoerd, hierbij kan de polymerisatie uitgevoerd worden door de toegevoegde DNA-polymerase. Afhankelijk van de grote van het fragment en de snelheid van de DNA-polymerase kan de laatste stap bij qPCR veelal worden overgeslagen, omdat de temperatuurwisseling tussen de annealing-stap en de denaturatie-stap voldoende is voor de DNA-polymerase om zijn werk volledig te doen. Tevens bestaat er ook een vierstaps qPCR waarbij er een extra stap wordt toegevoegd van enkele seconden bij bijvoorbeeld 80°C waarin de fluorescentie wordt gemeten, deze stap wordt toegevoegd om het signaal van primer-dimers (het aspecifiek binden van primers) te reduceren.[5] De exacte temperatuur en tijdsduur van iedere stap is afhankelijk van een groot aantal variabelen, waaronder de lengte van het DNA-fragment, de snelheid van het gekozen DNA-polymerase enzym en de annealingtemperatuur van de primers.[6]
Methodes[bewerken | brontekst bewerken]
De qPCR kan worden uitgevoerd op twee verschillende methodes, aspecifieke en specifieke detectie, beiden een ander doel en resultaat.
Aspecifieke detectie: qPCR met fluoroforen die binden aan dubbelstrengig DNA[bewerken | brontekst bewerken]
Bij aspecifieke detectie wordt een fluorofoor gebruikt die bind aan al het dubbelstrengig (ds) DNA. Doordat de hoeveelheid dsDNA na iedere PCR cyclus toeneemt, zal ook de fluorescentie van het sample toenemen na iedere cyclus. Het voordeel van aspecifieke detectie is dat het relatief eenvoudig en goedkoop is in zowel gebruik als opzet. Dit voordeel is gelijktijdig ook een groot nadeel, omdat aspecifieke fluoroforen (zoals bijvoorbeeld SYBR Green) bindt aan al het dsDNA zal het ook binden aan aspecifieke producten, zoals primer dimers. Dit kan mogelijk zorgen voor verstoringen in de metingen en leiden tot onbetrouwbare resultaten. qPCR reactiemengsels met aspecifieke detectie wordt in de basis op de zelfde wijze bereidt als een conventionele PCR reactiemengsel, er wordt enkel een fluorofoor aan toegevoegd.
Specifieke detectie: qPCR met fluoroforen die beschiken over een reporter probe[bewerken | brontekst bewerken]
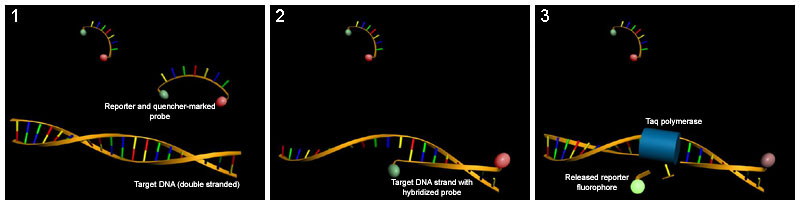
Bij specifieke detectie wordt een DNA probe gebruikt met daaraan een fluoroforen reporter. Door het gebruik van deze DNA probe zal er alleen fluorescentie ontstaan indien de probe specifiek heeft gebonden het het target-DNA. Hierdoor is deze methode significant meer specifiek dan aspecifieke detectie en maakt het mogelijk om zelfs bij de aanwezigheid van aspecifieke producten betrouwbare resultaten te krijgen. Indien er gebruik gemaakt wordt van probes met verschillende kleuren is het zelfs mogelijk om meerdere targets te monitoren binnen één sample. Dankzij specifieke detectie geven primer dimers niet langer gemeten omdat ze geen fluorescent-signaal kunnen geven, echter specifieke detectie voorkomt de vorming van primer dimers niet.
De methode werkt door middel van een DNA-gebaseerde probe met aan de ene zijde een fluorescente reporter en aan de andere zijde een verstoringsbron (quencher). Zolang deze reporter en quencher dicht bij elkaar zitten, zal er geen meetbaar fluorescent signaal worden geëxciteerd, echter op het moment dat de Taq polymerase de probe afbreekt tijdens de elongatie zal de reporter vrijkomen van de quencer waardoor het in staat is een een meetbaar fluorescent signaal uit te exciteren.
Smelttemperatuur analyse[bewerken | brontekst bewerken]

qPCR bied de mogelijkheid om verschillende geamplificeerde producten te detecteren door gebruik te maken van de smelttemperatuur van het DNA product (ook wel de Tm genoemd, naar het Engelse melting temperature). Deze techniek maakt gebruik van het feit dat bij aspecifieke detectie de fluorofoor bindt aan het dsDNA. Door de temperatuur langzaam te verhogen zal, zodra de Tm bereikt is, de hoeveelheid dsDNA snel afnemen waardoor ook het gemeten fluorescentie signaal afneemt. Doordat iedere DNA sequentie zijn eigen Tm heeft zal ook ieder product zijn eigen smeltcurve hebben. Hierdoor zijn bijvoorbeeld primer dimers relatief eenvoudig te detecteren, doordat deze een afwijking vormen op de smeltcurve.
Dataverwerking[bewerken | brontekst bewerken]
Een veelgebruikte methode om de hoeveelheid DNA te kwantificeren bij qPCR is door de gemeten excitatie te plotten op een logaritmische schaal waarbij gekeken wordt in welke cyclus het signaal een bepaalde drempelwaarde passeert. Doordat tegenstelling tot een conventionele PCR kan bij een qPCR tijdens het gehele proces de amplificatie gemonitord worden (vandaar dat qPCR ook wel Real-Time PCR wordt genoemd), is exact bekent hoeveel fluorescentie er in iedere cyclus is. De drempelwaarde wordt vastgesteld op 3 tot 5 maal de standaardafwijking van de achtergrondruis. Het nummer van de cyclus waarin het signaal de drempelwaarde passeert wordt ook wel de Ct-waarde genoemd (naar het Engelse threshold cycle).[8]
Gedurende de exponentiële amplificatie-fase verdubbeld de hoeveelheid DNA template theoretisch gezien iedere cyclus. Dat betekend dat als de Ct-waarde van sample A 3 cycli hoger is dan sample B, dat er in sample A theoretisch gezien 23 = 8 keer meer DNA zit dan in sample B. Echter in praktijk kan de daadwerkelijke efficiëntie hoger of lager liggen als gevolg van primer-template combinatie.
Om de genexpressie (ookwel Ratio of Fold difference) van een Gene Of Intrest (GOI) in zowel een RNA als een DNA sample te kunnen kwantificeren is normalisatie van de verkregen qPCR data noodzakelijk. In deze normalisatie wordt zowel voor achtergrond ruis gecorrigeerd, door het te vergelijken met een 'leeg' sample (een z.g.n. blanco of NTC (Negative Template Control), als wel voor celactiviteit, door het te vergelijken met het z.g.n. housekeeper-gen, welk in het zelfde sample aanwezig was.. Indien bekend, kan er ook voor de efficiëntie (E) van de reactie gecorrigeerd worden, wat de betrouwbaarheid van de data nog verder vergroot.[9]
- Indien de efficiëntie niet bekend is wordt gebruik gemaakt van de Livak-methode:
- R=2(ΔCT[Sample] - ΔCT[Housekeeper])
- Indien de efficiëntie wel bekend is wordt gebruik gemaakt van de Pfaffl-methode:
To quantify gene expression, the (Cq) for an RNA or DNA from the gene of interest is subtracted from the (Cq) of RNA/DNA from a housekeeping gene in the same sample to normalize for variation in the amount and quality of RNA between different samples. This normalization procedure is commonly called the ΔCt-method[10] and permits comparison of expression of a gene of interest among different samples. However, for such comparison, expression of the normalizing reference gene needs to be very similar across all the samples. Choosing a reference gene fulfilling this criterion is therefore of high importance, and often challenging, because only very few genes show equal levels of expression across a range of different conditions or tissues.[11][12] Although cycle threshold analysis is integrated with many commercial software systems, there are more accurate and reliable methods of analysing amplification profile data that should be considered in cases where reproducibility is a concern.
Mechanism-based qPCR quantification methods have also been suggested, and have the advantage that they do not require a standard curve for quantification. Methods such as MAK2[13] have been shown to have equal or better quantitative performance to standard curve methods. These mechanism-based methods use knowledge about the polymerase amplification process to generate estimates of the original sample concentration. An extension of this approach includes an accurate model of the entire PCR reaction profile, which allows for the use of high signal-to-noise data and the ability to validate data quality prior to analysis.
According to research of Ruijter et al.[14] MAK2 assumes constant amplification efficiency during the PCR reaction. However, theoretical analysis of polymerase chain reaction, from which MAK2 was derived, has revealed that amplification efficiency is not constant throughout PCR. While MAK2 quantification provides reliable estimates of target DNA concentration in a sample under normal qPCR conditions, MAK2 does not reliably quantify target concentration for qPCR assays with competimeters.
Applications[bewerken | brontekst bewerken]
There are numerous applications for quantitative polymerase chain reaction in the laboratory. It is commonly used for both diagnostic and basic research. Uses of the technique in industry include the quantification of microbial load in foods or on vegetable matter, the detection of GMOs (Genetically modified organisms) and the quantification and genotyping of human viral pathogens.
Diagnostic uses[bewerken | brontekst bewerken]
Diagnostic qualitative PCR is applied to rapidly detect nucleic acids that are diagnostic of, for example, infectious diseases, cancer and genetic abnormalities. The introduction of qualitative PCR assays to the clinical microbiology laboratory has significantly improved the diagnosis of infectious diseases,[15] and is deployed as a tool to detect newly emerging diseases, such as new strains of flu and coronavirus,[16] in diagnostic tests.[17][18]
Quantification of gene expression[bewerken | brontekst bewerken]
Quantifying gene expression by traditional DNA detection methods is unreliable. Detection of mRNA on a northern blot or PCR products on a gel or Southern blot does not allow precise quantification.[19] For example, over the 20–40 cycles of a typical PCR, the amount of DNA product reaches a plateau that is not directly correlated with the amount of target DNA in the initial PCR.[20]
Real-time PCR can be used to quantify nucleic acids by two common methods: relative quantification and absolute quantification.[21] Absolute quantification gives the exact number of target DNA molecules by comparison with DNA standards using a calibration curve. It is therefore essential that the PCR of the sample and the standard have the same amplification efficiency.[22] Relative quantification is based on internal reference genes to determine fold-differences in expression of the target gene. The quantification is expressed as the change in expression levels of mRNA interpreted as complementary DNA (cDNA, generated by reverse transcription of mRNA). Relative quantification is easier to carry out as it does not require a calibration curve as the amount of the studied gene is compared to the amount of a control reference gene.
As the units used to express the results of relative quantification are unimportant the results can be compared across a number of different RTqPCR. The reason for using one or more housekeeping genes is to correct non-specific variation, such as the differences in the quantity and quality of RNA used, which can affect the efficiency of reverse transcription and therefore that of the whole PCR process. However, the most crucial aspect of the process is that the reference gene must be stable.[23]
The selection of these reference genes was traditionally carried out in molecular biology using qualitative or semi-quantitative studies such as the visual examination of RNA gels, northern blot densitometry or semi-quantitative PCR (PCR mimics). Now, in the genome era, it is possible to carry out a more detailed estimate for many organisms using transcriptomic technologies.[24] However, research has shown that amplification of the majority of reference genes used in quantifying the expression of mRNA varies according to experimental conditions.[25][26][27] It is therefore necessary to carry out an initial statistically sound methodological study in order to select the most suitable reference gene.
A number of statistical algorithms have been developed that can detect which gene or genes are most suitable for use under given conditions. Those like geNORM or BestKeeper can compare pairs or geometric means for a matrix of different reference genes and tissues.[28][29]
Microbiological uses[bewerken | brontekst bewerken]
Quantitative PCR is also used by microbiologists working in the fields of food safety, food spoilage and fermentation and for the microbial risk assessment of water quality (drinking and recreational waters) and in public health protection.[30]
qPCR may also be used to amplify taxonomic or functional markers of genes in DNA taken from environmental samples.[31] Markers are represented by genetic fragments of DNA or complementary DNA.[31] By amplifying a certain gentic element, one can quantify the amount of the element in the sample prior to amplification.[31] Using taxonomic markers (ribosomal genes) and qPCR can help determine the amount of microorganisms in a sample, and can identify different families, genera, or species based on the specificity of the marker.[31] Using functional markers (protein-coding genes) can show gene expression within a community, which may reveal information about the environment.[31]
Detection of phytopathogens[bewerken | brontekst bewerken]
The agricultural industry is constantly striving to produce plant propagules or seedlings that are free of pathogens in order to prevent economic losses and safeguard health. Systems have been developed that allow detection of small amounts of the DNA of Phytophthora ramorum, an oomycete that kills Oaks and other species, mixed in with the DNA of the host plant. Discrimination between the DNA of the pathogen and the plant is based on the amplification of ITS sequences, spacers located in ribosomal RNA gene's coding area, which are characteristic for each taxon.[32] Field-based versions of this technique have also been developed for identifying the same pathogen.[33]
Detection of genetically modified organisms[bewerken | brontekst bewerken]
qPCR using reverse transcription (RT-qPCR) can be used to detect GMOs given its sensitivity and dynamic range in detecting DNA. Alternatives such as DNA or protein analysis are usually less sensitive. Specific primers are used that amplify not the transgene but the promoter, terminator or even intermediate sequences used during the process of engineering the vector. As the process of creating a transgenic plant normally leads to the insertion of more than one copy of the transgene its quantity is also commonly assessed. This is often carried out by relative quantification using a control gene from the treated species that is only present as a single copy.[34][35]
Clinical quantification and genotyping[bewerken | brontekst bewerken]
Viruses can be present in humans due to direct infection or co-infections which makes diagnosis difficult using classical techniques and can result in an incorrect prognosis and treatment. The use of qPCR allows both the quantification and genotyping (characterization of the strain, carried out using melting curves) of a virus such as the Hepatitis B virus.[36] The degree of infection, quantified as the copies of the viral genome per unit of the patient's tissue, is relevant in many cases; for example, the probability that the type 1 herpes simplex virus reactivates is related to the number of infected neurons in the ganglia.[37] This quantification is carried out either with reverse transcription or without it, as occurs if the virus becomes integrated in the human genome at any point in its cycle, such as happens in the case of HPV (human papillomavirus), where some of its variants are associated with the appearance of cervical cancer.[38]
|
Bron
Referenties
Bibliografie
|

![{\displaystyle R={\frac {E_{[sample]}^{(\triangle C_{T[sample]})}}{E_{[housekeeper]}^{(\triangle C_{T[housekeper]})}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/e2f87fb5576de7916fbe19993b220ea529659a96)