Secundaire structuur (nucleïnezuur)


De secundaire structuur van een nucleïnezuur is de manier waarop de nucleotideketen met zichzelf of met een andere nucleotideketen verbonden is via basenparing. DNA- en RNA-moleculen hebben zeer verschillende secundaire structuren: DNA is dubbelstrengs en bestaat uit een continue dubbele helix, RNA bevat vaak zelfcomplementaire sequenties die zorgen dat het RNA met zichzelf kan basenparen waardoor het in lokale elementen wordt opgevouwen.
De secundaire structuur komt bij biologische moleculen altijd tot stand via waterstofbruggen. In een niet-biologische context is de secundaire structuur een belangrijk aspect bij het ontwerpen van nucleïnezuurstructuren voor DNA-nanotechnologie. Het patroon van de basenparen bepaalt de secundaire structuur en uiteindelijk de algehele ruimtelijke vorm van een nucleïnezuur. Een aantal belangrijke secundaire structuurmotieven zijn de haarspeldlus en de pseudoknoop.
Algemeen[bewerken | brontekst bewerken]
Basenparing[bewerken | brontekst bewerken]
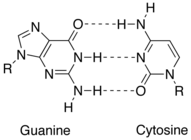
Wanneer twee nucleotiden op tegenover elkaar liggende complementaire DNA- of RNA-strengen waterstofbruggen vormen, noemt men dit een basenpaar. In de conventionele Watson-Crick-basenparing vormt adenine (A) een basenpaar met thymine (T) en vormt guanine (G) er een met cytosine (C). In RNA wordt thymine vervangen door uracil (U). Doordat een keten van nucleotiden kan fungeren als 'matrijs' voor de aanmaak van een complementaire keten nucleotiden, kan genetische informatie tijdens replicatie gericht in stand blijven. Basenparing is bovendien het mechanisme waarmee codons op messenger-RNA herkend worden door anticodons op transfer-RNA tijdens de translatie. Naast replicatie is basenparing is dus ook essentieel voor de eiwitsynthese.
Basenparing is mogelijk door de vorming van waterstofbruggen tussen de basen. Bij een enkelstrengse DNA-keten vormen de basen vrij sterke waterstofbruggen met watermoleculen. Pas wanneer de dubbele helix zich vormt, ontstaan er waterstofbruggen tussen de nucleotiden. In tegenstelling tot wat vaak wordt aangenomen spelen de waterstofbruggen geen grote rol bij het stabiliseren van de DNA-structuur. Hiervoor zijn voornamelijk stacking interactions tussen de basenparen verantwoordelijk.[1] Stacking is de aantrekkingskracht tussen aromatische ringen (in dit geval de basen), en is met name tussen GC-basenparen zeer sterk. Om deze reden is DNA met een hoog GC-gehalte per definitie stabieler dan DNA met een laag GC-gehalte.[2]
Adenine en guanine behoren tot een klasse van bicyclische verbindingen genaamd purines. De kleinere nucleobasen cytosine en thymine (en uracil) behoren tot een klasse van monocyclische verbindingen die pyrimidines worden genoemd. Purines zijn complementair aan pyrimidines: een pyrimidine-pyrimidine-paar is energetisch ongunstig omdat de moleculen te ver uit elkaar liggen om waterstofbruggen te vormen; een purine-purine-paar is energetisch ongunstig omdat de moleculen te dicht bij elkaar liggen, wat leidt tot afstoting.
Denaturatie en renaturatie[bewerken | brontekst bewerken]
Wanneer een waterige oplossing van DNA wordt verwarmd tot 90–100 °C of wordt blootgesteld aan een extreem hoge pH, dan zullen de waterstofbruggen tussen de complementaire ketens loslaten.[3] Het DNA splitst uiteen in twee afzonderlijke strengen. Dit proces wordt denaturatie genoemd. Wanneer de oplossing wordt afgekoeld zullen de complementaire strengen zich weer samenvoegen tot een dubbelstrengse helixstructuur. Men spreekt dan van renaturatie of hybridisatie. Wanneer een kort stukje DNA of RNA renatureert, bijvoorbeeld een synthetische oligonucleotide die gebruikt wordt als primer, spreekt men van annealing.[3]
Het uit elkaar gaan van een dubbelstrengs nucleïnezuur gebeurt niet overal in het nucleïnezuur even gemakkelijk.[4] AT-rijke sequenties denatureren sneller dan GC-rijke sequenties, doordat adenine en thymine verbonden zijn door slechts twee waterstofbruggen. Dit is de reden dat er aan het begin van een eukaryotisch gen vaak een TATAA-sequentie voorkomt: het DNA gaat hier gemakkelijk uit elkaar zodat RNA-polymerase de naliggende structuurgenen kan aflezen.
DNA denatureren door het te verwarmen, zoals in een PCR, is alleen mogelijk als het DNA-molecuul uit minder dan ongeveer 10.000 basenparen bestaat. Een langer DNA-molecuul is vaak dermate in zichzelf verstrengeld, dat uiteensplitsing onmogelijk is. In een levende cel is een enzym aanwezig dat deze verstrengelingen lokaal oplost: topo-isomerase. Dit enzym kan de bindingen in de ruggengraat van de nucleotidestrengen verbreken en vernieuwen. Door denaturerende enzymen (helicases) gelijktijdig te laten werken met topo-isomerases, kan een volledig chromosoom gerepliceerd worden.[5]
Motieven[bewerken | brontekst bewerken]
De secundaire structuur van nucleïnezuren is zeer divers. Er zijn meerdere elementen of motieven van de secundaire structuur te onderscheiden: helices (die optreden bij aaneengesloten basenparen) en verschillende lussen (die optreden bij ongepaarde nucleotiden). Belangrijke voorbeelden van lussen zijn de haarspeldlus, de pseudoknoop en de tetralus. Lussen komen voor bij RNA-moleculen. De secundaire structuurmotieven vormen uiteindelijk samen een complexe tertiaire structuur.
Dubbele helix[bewerken | brontekst bewerken]

De dubbele helix is veruit de belangrijkste ruimtelijke configuratie van een nucleïnezuur: DNA neemt onder normale omstandigheden altijd deze structuur aan. De dubbele helix komt rechtstreeks voort uit de secundaire structuur van het molecuul, het patroon van basenparing. Er bestaan voor DNA drie biologisch relevante helixstructuren: A-DNA, B-DNA en Z-DNA. Dit zijn drie verschillende vormen van de dubbele helix (tertiaire structuur), maar bij elk is het patroon van basenparing hetzelfde (secundaire structuur).
Bij nucleïnezuren is de dubbele helix meestal een rechtsdraaiend polymeer bestaande uit twee nucleotideketens die om elkaar heen zijn gevlochten, waarbij de nucleotiden van iedere keten verbonden zijn als de treden van een wenteltrap. Een enkele draai van de helix bestaat uit ongeveer tien nucleotiden en kent een brede groeve en een smalle groeve. De brede groeve is de plaats waar eiwitten (zoals transcriptiefactoren) aan het DNA kunnen binden, omdat hier voldoende ruimte is om interacties aan te gaan met de nucleotiden.[6]
Haarspeldlus[bewerken | brontekst bewerken]

De haarspeldlus (Engels: hairpin-loop of stem-loop) is een ander belangrijk motief van de secundaire structuur. Een haarspeldlus is te beschouwen als een enkelstrengse RNA-keten die dubbelgevouwen is, zodat er een korte lus met een aantal ongepaarde nucleotiden ontstaat. De 'stam' wordt gestabiliseerd door zelfcomplementaire basenparing. Haarspeldlussen komen zeer veel voor in RNA-moleculen, bijvoorbeeld in de klaverbladstructuur van transfer-RNA.
Een haarspeldlus is vaak van groot belang voor de functionaliteit van een RNA-molecuul; naast transfer-RNA is de Rho-onafhankelijke terminator een bekend voorbeeld. Er is veel onderzoek gedaan om de secundaire structuur van RNA-moleculen te achterhalen. Zowel experimentele als computationele benaderingen worden gebruikt.[7][8]
Pseudoknoop[bewerken | brontekst bewerken]

Een pseudoknoop (Engels: pseudoknot) is een secundaire structuurmotief dat uit twee of meer haarspeldlussen bestaat, waarbij de ‘stam’ van de ene haarspeldlus gedeeltelijk geïntercaleerd is in de stam van de andere haarspeldlus: de lus van haarspeldlus A gaat basenparen met de stam van haarspeldlus B. Pseudoknopen danken hun naam aan de knoopvormige driedimensionale conformaties waarin ze zich organiseren, maar het zijn geen werkelijke topologische knopen. Doordat de basenparen in een pseudoknoop overlappen in elkaar vervlochten zijn, zijn ze zeer moeilijk te voorspellen uit de nucleotidesequentie.[10]
Veel pseudoknopen vertonen een bepaalde katalytische activiteit.[11] Er bestaan verschillende RNA-moleculen die voor hun biologische functie een pseudoknoop nodig hebben, bijvoorbeeld het RNA-component van menselijk telomerase. Hoewel pseudoknopen ook in DNA kunnen ontstaan, zijn ze over het algemeen niet aanwezig onder fysiologische omstandigheden.
Zie ook[bewerken | brontekst bewerken]
Referenties
Bronnen
|