Experimentele evolutie
Experimentele evolutie is het gebruik van laboratoriumexperimenten of gecontroleerde veldproeven om de evolutionaire dynamiek te onderzoeken.[1] Evolutie kan in het laboratorium worden waargenomen als individuen/populaties zich aanpassen aan nieuwe omgevingsomstandigheden door natuurlijke selectie.
Er zijn twee verschillende manieren waarop aanpassing kan ontstaan in de experimentele evolutie. Eén daarvan is dat een individueel organisme een nieuwe gunstige mutatie verkrijgt..[2] De andere is een allel frequentieverandering in de genetische variatie die al aanwezig is in een populatie van organismen.[2] Andere evolutionaire krachten buiten mutatie en natuurlijke selectie kunnen ook een rol spelen of worden opgenomen in experimentele evolutiestudies, zoals genetische drift en gene flow.[3]
Welk organisme wordt gebruikt, wordt bepaald door de onderzoeker, op basis van de te testen hypothese. Er zijn veel generaties nodig om adaptieve mutatie te laten plaatsvinden, waarom experimentele evolutie via mutatie wordt uitgevoerd in virussen of eencelligen met snelle generatietijden, zoals bacteriën en aseksuele klonale gist.[1][4][5] Polymorfe populaties van aseksuele of seksuele gist,[2] en meercellige eukaryoten zoals Drosophila kunnen zich aanpassen aan nieuwe omgevingen door allelfrequentieverandering in bestaande genetische variatie.[3] Organismen met langere generatietijden kunnen, hoewel kostbaar, worden gebruikt in experimentele evolutie. Laboratoriumstudies met vossen[6] en met knaagdieren (zie hieronder) hebben aangetoond dat opmerkelijke aanpassingen kunnen plaatsvinden binnen slechts 10-20 generaties, ook experimenten met wilde guppies hebben aanpassingen waargenomen binnen vergelijkbare aantallen generaties.[7]
Meer recentelijk worden experimenteel ontwikkelde individuen of populaties vaak geanalyseerd door het hele genoom te sequencen,[8][9] een benadering die bekend staat als beoordeel de verandering en sequence opnieuw (E&R-Evolve and Resequence).[10] E&R kan mutaties identificeren die leiden tot aanpassing bij klonale individuen of allelen identificeren die in frequentie veranderden in polymorfe populaties, door de sequenties van individuen/populaties voor en na aanpassing te vergelijken.[2] De sequentiegegevens maken het mogelijk om de plaats in een DNA-sequentie te lokaliseren waar een mutatie/allelfrequentieverandering heeft plaatsgevonden, die de aanpassing tot stand heeft gebracht.[10][9][2] De aard van de aanpassing en functionele vervolgstudies kunnen inzicht geven in welk effect de mutatie/allel heeft op fenotype.
Geschiedenis[bewerken | brontekst bewerken]
Domesticatie en fokken[bewerken | brontekst bewerken]


Onbewust hebben mensen evolutie-experimenten uitgevoerd zolang ze planten en dieren gedomesticeerd hebben. Veredeling van planten en fokken van dieren heeft geleid tot rassen en variëteiten die dramatisch verschillen van hun oorspronkelijke wildtype-voorouders. Voorbeelden zijn de koolsoorten, banaan, maïs, of het grote aantal verschillende hondenrassen. Het fokken van rassen en variëteiten door de mens die extreme verschillen vertonen in één enkele soort werd al erkend door Charles Darwin. In feite begon hij zijn boek The Origin of Species met een hoofdstuk over de variatie bij huisdieren. In dit hoofdstuk besprak Darwin vooral de duif.
Vroegste experimenten[bewerken | brontekst bewerken]

Een van de eersten die een gecontroleerd evolutie-experiment uitvoerde was William Dallinger. Aan het einde van de 19e eeuw kweekte hij gedurende een periode van zeven jaar (1880-1886) kleine eencelligen in een op maat gemaakte broedmachine. Dallinger verhoogde langzaam de temperatuur van de broedmachine van aanvankelijk 60°F tot 158°F. De vroege culturen vertoonden duidelijke tekenen van angst bij een temperatuur van 73°F, en waren zeker niet in staat om te overleven bij 158°F. De organismen die Dallinger aan het einde van het experiment in zijn incubator had, waren daarentegen prima in orde bij 158 ° F. Deze organismen zouden echter niet langer groeien bij de aanvankelijke 60°F. Dallinger concludeerde dat hij in zijn broedmachine bewijs had gevonden voor de darwinistische aanpassing en dat de organismen zich hadden aangepast om in een omgeving met hoge temperaturen te kunnen leven. De broedmachine van Dallinger werd per ongeluk vernietigd in 1886 en Dallinger kon deze onderzoekslijn niet voortzetten.[11][12]
In de jaren 1880 tot 1980 werd experimentele evolutie met tussenpozen beoefend door een verscheidenheid aan evolutiebiologen, waaronder de zeer invloedrijke Theodosius Dobzhansky. Net als bij ander experimenteel onderzoek in de evolutiebiologie in deze periode ontbrak het bij veel van dit werk aan uitgebreide herhalingen en werd het slechts gedurende relatief korte perioden van de evolutionaire tijd uitgevoerd.[13]
Latere experimenten[bewerken | brontekst bewerken]
Experimentele evolutie is in verschillende experimenten uitgevoerd om onderliggende evolutionaire processen in een gecontroleerd systeem te begrijpen. Er is experimentele evolutie uitgevoerd op meercelligen[14] en eencelligen[15] eukaryoten, prokaryoten,[16] en virussen.[17] Vergelijkbare proeven zijn ook uitgevoerd door gerichtete evolutie van individuele enzymen,,[18][19] ribozymen[20] en zelfreplicerende[21][22] genen.
Bladluizen[bewerken | brontekst bewerken]
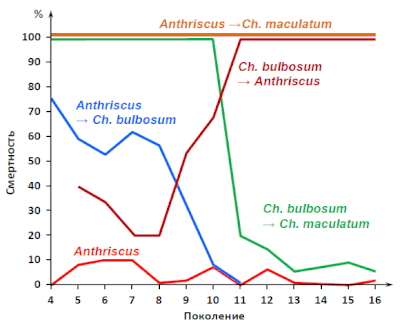
In de jaren vijftig voerde de Sovjet-bioloog Georgy Shaposhnikov experimenten uit op bladluizen van het geslacht Dysaphis. In het experiment met D. antkrisci rnajkopica Shap. (fluitenkruidluis) zijn afstammelingen van één oorspronkelijk individu – de stichter – sinds mei opgegroeid op hun secundaire gastheer (Anthriscus nemorosa). Een ander deel van de nakomelingen van hetzelfde oorspronkelijke individu is acht generaties lang opgegroeid op een plant waar ze weinig aan hadden (Chaerophyllum bulbosum). Door het proces van intensieve natuurlijke selectie hebben de bladluizen zich volledig aangepast aan de nieuwe plantensoort. Tegelijkertijd verloren ze het vermogen om op hun vorige gastheer (Anthriscus nemorosa) te leven en verwierven ze het vermogen om zich aan te passen aan een voorheen ongeschikte plant (Chaerophyllum maculatum), waarop ze vervolgens normaal leefden en zich vele generaties lang voortplanten. Als gevolg van divergentie ontstond een nieuwe soort. Het verwierf statistisch significante morfologische verschillen met de oorspronkelijke vorm en verloor tegelijkertijd enkele van de belangrijkste verschillen met de nauw verwante vorm D. chaerophyllina[23]
Fruitvliegjes[bewerken | brontekst bewerken]
Een van de eerste van een nieuwe golf van experimenten waarbij deze strategie werd gebruikt, was de laboratorium-"evolutionaire straling" van Drosophila melanogaster-populaties waarmee Michael R. Rose in februari 1980 begon.[24] Dit systeem begon met tien populaties, waarvan er vijf op latere leeftijd werden gekweekt en vijf op jonge leeftijd werden gekweekt. Sindsdien zijn er met deze laboratoriumstraling meer dan 200 verschillende populaties gecreëerd, waarbij de selectie zich op meerdere karakters richtte. Sommige van deze sterk gedifferentieerde populaties zijn ook ‘achteruit’ of ‘omgekeerd’ geselecteerd, door experimentele populaties terug te brengen naar hun voorouderlijk cultuurregime. Honderden mensen hebben gedurende het grootste deel van drie decennia met deze populaties gewerkt. Veel van dit werk is samengevat in de artikelen verzameld in het boek Methuselah Flies.[25]
De eerste experimenten met de vliegen waren beperkt tot het bestuderen van fenotypen, maar de moleculaire mechanismen, dat wil zeggen de veranderingen in het DNA die dergelijke veranderingen mogelijk maakten, konden niet worden geïdentificeerd. Dit veranderde met de genomics-technologie.[26] Vervolgens bedacht Thomas Turner de term Evolve and Resequence (E&R).[10] Verschillende onderzoeken gebruikten de E&R-aanpak met wisselend succes.[27][28] Een van de interessantere experimentele evolutiestudies werd uitgevoerd door de groep van Gabriel Haddad aan de UC San Diego, waar Haddad en collega's vliegen ontwikkelden voor het aanpassen aan omgevingen met weinig zuurstof, ook wel bekend als hypoxie.[29] Na 200 generaties gebruikten ze de E&R-aanpak om genomische regio's te identificeren die door natuurlijke selectie waren geselecteerd in de aan hypoxie aangepaste vliegen.[30] Recentere experimenten volgen E&R-voorspellingen op met RNAsequencing[31] en genetische kruisingen.[9] Dergelijke inspanningen bij het combineren van E&R met experimentele validaties zouden krachtig moeten zijn bij het identificeren van genen die de aanpassing bij vliegen reguleren.
Micro-organismen[bewerken | brontekst bewerken]
Veel micro-organismen hebben een korte generatietijd, hebben gemakkelijk te sequencen genomen en een goed begrepen biologie. Ze worden daarom vaak gebruikt voor experimentele evolutiestudies. De bacteriesoorten die het meest worden gebruikt voor experimentele evolutie zijn onder meer Pseudomonas fluorescens,[32] Pseudomonas aeruginosa,[33] Enterococcus faecalis[34] en Escherichia coli (zie hieronder), terwijl de gist Saccharomyces cerevisiae is gebruikt als model voor de studie van de eukaryotische evolutie.[35]
Lenski's Escherichia coli experiment[bewerken | brontekst bewerken]
Eén van de meest bekende voorbeelden van bacteriële evolutie in het laboratorium is het E. coli langetermijnevolutie-experiment van Richard Lenski. Op 24 februari 1988 begon Lenski met het kweken van twaalf stammen van E. coli onder identieke groeiomstandigheden.[36][37] Toen een van de populaties het vermogen ontwikkelde om citroenzuur uit het groeimedium aeroob te metaboliseren en een sterk toegenomen groei vertoonde,[38] leverde dit een dramatische observatie op van evolutie in actie. Het experiment gaat tot op de dag van vandaag door en is nu het langstlopende (in termen van generaties) gecontroleerde evolutie-experiment dat ooit is uitgevoerd. Sinds het begin van het experiment zijn er meer dan 60.000 generaties verlopen. Lenski en collega's publiceren regelmatig updates over de status van de experimenten.[39]
Leishmania donovani[bewerken | brontekst bewerken]
Bussotti en medewerkers isoleerden amastigoten uit Leishmania donovani, een trypanosome protozoa en kweekten ze in vitro gedurende 3800 generaties (36 weken). De kweek van deze parasieten liet zien hoe ze zich aanpasten aan in vitro omstandigheden door het verlies van een NIMA-gerelateerde kinase 1 te compenseren, belangrijk voor de correcte voortgang van de mitose, door de expressie van een andere homologe kinase te verhogen naarmate de kweekgeneraties vorderden. Verder werd waargenomen hoe L. donovani is aangepast aan in vitro cultuur door de expressie van 23 transcripten gerelateerd aan flagelle biogenese te verminderen en de expressie van ribosomale eiwitclusters en niet-coderende RNA's zoals nucleolar small RNA's. De flagel wordt door de parasiet in de in vitro cultuur als minder noodzakelijk beschouwd en daarom leidt de progressie van generaties tot de eliminatie ervan, waardoor een energiebesparing ontstaat als gevolg van de lagere motiliteit, zodat de proliferatie en de groeisnelheid in de cultuur hoger zijn. De geamplificeerde snoRNA's leiden ook tot verhoogde ribosomale biosynthese, verhoogde eiwitbiosynthese en dus tot verhoogde groeisnelheid van de cultuur. Deze aanpassingen die gedurende generaties parasieten worden waargenomen, worden bepaald door copynumbervariaties (CNV) en epistatische interacties tussen betrokken genen en stellen ons in staat de genomische instabiliteit van Leishmania door de post-transcriptionele regulatie van genexpressie aan te tonen.[40]
Laboratoriumhuismuizen[bewerken | brontekst bewerken]

In 1998 startten Theodore Garland, Jr. en collega's een langetermijnexperiment met het selectief fokken van laboratoriumhuismuizen voor hoge vrijwillige activiteitsniveaus in tredmolens.[41] Dit experiment gaat ook tot op de dag van vandaag door (> 90 generaties). Muizen van de vier "High Runner"-lijnen (HR-lijnen) zijn geëvolueerd om bijna drie keer zoveel tredmolenomwentelingen per dag te draaien vergeleken met de vier niet-geselecteerde controlelijnen van muizen, voornamelijk door sneller te rennen dan de controlemuizen in plaats van meer minuten per dag te rennen.

De HR-muizen vertonen een verhoogde maximale zuurstofopnamevermogen wanneer ze worden getest op een gemotoriseerde loopband. Ze vertonen ook veranderingen in motivatie en het beloningssysteem van de hersenen. Farmacologische onderzoeken wijzen op veranderingen in de functie van dopamine en het endocannabinoïdesysteem.[42] De HR-lijnen zijn voorgesteld als een model om de menselijke aandachtstekort-hyperactiviteitstoornis (ADHD) te bestuderen. De toediening van Ritalin bleek hun tredloopsnelheid te verminderen ongeveer tot de niveaus van de controlemuizen.
Multidirectionele selectie op rosse woelmuizen[bewerken | brontekst bewerken]
In 2005 startte Paweł Koteja samen met Edyta Sadowska en collega's van de Jagiellonische Universiteit (Polen) een multidirectionele selectie op een niet-laboratoriumknaagdier, de rosse woelmuis Myodes (= Clethrionomys) glareolus.[43] De rosse woelmuizen werden geselecteerd op drie verschillende eigenschappen, die een belangrijke rol speelden in de adaptieve radiatie van gewervelde landdieren: hoge maximale snelheid van een aerobe metabolisme, neiging tot predatie en herbivoor vermogen. Aerobe lijnen werden geselecteerd op basis van het maximale zuurstofverbruik dat werd bereikt tijdens het zwemmen bij 38°C; Predatie-lijnen – voor een korte tijd voor het vangen van levende krekels; herbivore-lijnen – voor het vermogen om de lichaamsmassa te behouden wanneer ze een dieet van lage kwaliteit kregen, “verdund” met gedroogd graspoeder. Er werden vier nakomelingenlijnen aangehouden voor elk van de drie selectierichtingen en nog eens vier als niet-geselecteerde controles.
Na ongeveer 20 generaties selectief fokken ontwikkelden de rosse woelmuizen uit de aerobe lijnen een 60% hogere door zwemmen geïnduceerde stofwisseling dan woelmuizen uit de niet-geselecteerde controlelijnen. Hoewel het selectieprotocol geen thermoregulerende belasting oplegt, zijn zowel de basale stofwisseling als thermogene capaciteit toegenomen in de aerobe-lijnen.[44][45] De resultaten hebben dus enige ondersteuning geboden voor het “aerobe capaciteitsmodel” voor de evolutie van warmbloedigheid bij zoogdieren.
Meer dan 85% van de predatore rosse woelmuizen vangen de krekels, vergeleken met slechts ongeveer 15% van de niet-geselecteerde controle rosse woelmuizen en ze vangen de krekels sneller. Het toegenomen predatie gedrag gaat gepaard met een meer proactieve coping-stijl (“persoonlijkheid”).[46]
Tijdens de test met een dieet van lage kwaliteit verliezen de plantenetende rosse woelmuizen ongeveer 2 gram minder massa (ongeveer 10% van de oorspronkelijke lichaamsmassa) dan de controlemuizen. De herbivore rosse woelmuizen hebben een veranderde samenstelling van de bacteriële microbioom in hun blindedarm[47] De selectie heeft dus geresulteerd in de evolutie van de gehele holobiont en het experiment kan een laboratoriummodel bieden van de hologenome evolutie.
Synthetische biologie[bewerken | brontekst bewerken]
Synthetische biologie biedt unieke mogelijkheden voor experimentele evolutie, waarbij de interpretatie van evolutionaire veranderingen wordt vergemakkelijkt door genetische elementen in gastheergenomen in te voegen en selectie toe te passen die specifiek op dergelijke elementen is gericht. Synthetische biologische netwerken ingevoegd in het genoom van Escherichia coli[48] of de knopvormende gist Saccharomyces cerevisiae[49] degraderen (verliest functie) tijdens laboratoriumevolutie. Met de juiste selectie kunnen de mechanismen die ten grondslag liggen aan het evolutionaire herstel van een verloren biologische functie worden bestudeerd.[50] Experimentele evolutie van zoogdiercellen die synthetische gennetwerken bevatten[51] laten de rol van cellulaire heterogeniteit in de evolutie van resistentie tegen geneesmiddelen zien, met gevolgen voor de chemotherapieresistentie van kankercellen.
Andere voorbeelden[bewerken | brontekst bewerken]
Stekelbaarzen hebben zowel zee- als zoetwatersoorten, waarbij de zoetwatersoorten zich sinds de laatste ijstijd hebben ontwikkeld. Zoetwatersoorten kunnen koudere temperaturen overleven. Wetenschappers hebben getest of ze deze evolutie van koudetolerantie konden reproduceren door zee-stekelbaarsjes in koud zoet water te houden. Het kostte de zee-stekelbaarsjes slechts drie generaties om te evolueren naar een verbetering van 2,5 graden Celsius in koudetolerantie, die wordt aangetroffen bij wilde zoetwaterstekelbaarsjes.[52]
Microbiële cellen[53] en recentelijk zoogdiercellen (Chinese dwerghamster-eierstokcellen)[54] zijn ontwikkeld onder voedingsstoffen beperkende omstandigheden om hun metabolische respons te bestuderen en cellen te ontwikkelen voor nuttige eigenschappen.
Referenties[bewerken | brontekst bewerken]
- ↑ a b Experimental Evolution. Nature.
- ↑ a b c d e Long A, Liti G, Luptak A, Tenaillon O (October 2015). Elucidating the molecular architecture of adaptation via evolve and resequence experiments. Nature Reviews. Genetics 16 (10): 567–582. PMID 26347030. PMC 4733663. DOI: 10.1038/nrg3937.
- ↑ a b Kawecki TJ, Lenski RE, Ebert D, Hollis B, Olivieri I, Whitlock MC (oktober 2012). Experimentele evolutie. Trends in ecologie en evolutie 27: 547–560. PMID 22819306. DOI: 10.1016/j.tree.2012.06.001.
- ↑ Buckling A, Craig Maclean R, Brockhurst MA, Colegrave N (February 2009). The Beagle in a bottle. Nature 457 (7231): 824–829. PMID 19212400. DOI: 10.1038/nature07892.
- ↑ Elena SF, Lenski RE (June 2003). Evolution experiments with microorganisms: the dynamics and genetic bases of adaptation. Nature Reviews. Genetics 4 (6): 457–469. PMID 12776215. DOI: 10.1038/nrg1088.
- ↑ Trut LN (March 1999). Early Canid Domestication: The Farm-Fox Experiment: Foxes bred for tamability in a 40-year experiment exhibit remarkable transformations that suggest an interplay between behavioral genetics and development.. American Scientist 87 (2): 160–169. DOI: 10.1511/1999.2.160.
- ↑ Reznick DN, Shaw FH, Rodd FH, Shaw RG (March 1997). Evaluation of the Rate of Evolution in Natural Populations of Guppies (Poecilia reticulata). Science 275 (5308): 1934–1937. PMID 9072971. DOI: 10.1126/science.275.5308.1934.
- ↑ Barrick JE, Lenski RE (December 2013). Genome dynamics during experimental evolution. Nature Reviews. Genetics 14 (12): 827–839. PMID 24166031. PMC 4239992. DOI: 10.1038/nrg3564.
- ↑ a b c Jha AR, Miles CM, Lippert NR, Brown CD, White KP, Kreitman M (October 2015). Whole-Genome Resequencing of Experimental Populations Reveals Polygenic Basis of Egg-Size Variation in Drosophila melanogaster. Molecular Biology and Evolution 32 (10): 2616–2632. PMID 26044351. PMC 4576704. DOI: 10.1093/molbev/msv136.
- ↑ a b c Turner TL, Stewart AD, Fields AT, Rice WR, Tarone AM (March 2011). Population-based resequencing of experimentally evolved populations reveals the genetic basis of body size variation in Drosophila melanogaster. PLOS Genetics 7 (3): e1001336. PMID 21437274. PMC 3060078. DOI: 10.1371/journal.pgen.1001336.
- ↑ Haas JW (January 2000). The Reverend Dr William Henry Dallinger, F.R.S. (1839-1909). Notes and Records of the Royal Society of London 54 (1): 53–65. PMID 11624308. DOI: 10.1098/rsnr.2000.0096.
- ↑ Zimmer C (2011), Darwin Under the Microscope: Witnessing Evolution in Microbes. W. H. Freeman, 42–43. ISBN 978-0981519494.
- ↑ Dobzhansky T, Pavlovsky O (1957). An experimental study of interaction between genetic drift and natural selection. Evolution 11 (3): 311–319. DOI: 10.2307/2405795.
- ↑ Marden JH, Wolf MR, Weber KE (November 1997). Aerial performance of Drosophila melanogaster from populations selected for upwind flight ability. The Journal of Experimental Biology 200 (Pt 21): 2747–2755. PMID 9418031. DOI: 10.1242/jeb.200.21.2747.
- ↑ Ratcliff WC, Denison RF, Borrello M, Travisano M (January 2012). Experimental evolution of multicellularity. Proceedings of the National Academy of Sciences of the United States of America 109 (5): 1595–1600. PMID 22307617. PMC 3277146. DOI: 10.1073/pnas.1115323109.
- ↑ Barrick JE, Yu DS, Yoon SH, Jeong H, Oh TK, Schneider D, Lenski RE, Kim JF (October 2009). Genome evolution and adaptation in a long-term experiment with Escherichia coli. Nature 461 (7268): 1243–1247. PMID 19838166. DOI: 10.1038/nature08480.
- ↑ Heineman RH, Molineux IJ, Bull JJ (August 2005). Evolutionary robustness of an optimal phenotype: re-evolution of lysis in a bacteriophage deleted for its lysin gene. Journal of Molecular Evolution 61 (2): 181–191. PMID 16096681. DOI: 10.1007/s00239-004-0304-4.
- ↑ Bloom JD, Arnold FH (June 2009). In the light of directed evolution: pathways of adaptive protein evolution. Proceedings of the National Academy of Sciences of the United States of America 106 (Suppl 1): 9995–10000. PMID 19528653. PMC 2702793. DOI: 10.1073/pnas.0901522106.
- ↑ Moses AM, Davidson AR (May 2011). In vitro evolution goes deep. Proceedings of the National Academy of Sciences of the United States of America 108 (20): 8071–8072. PMID 21551096. PMC 3100951. DOI: 10.1073/pnas.1104843108.
- ↑ Salehi-Ashtiani K, Szostak JW (November 2001). In vitro evolution suggests multiple origins for the hammerhead ribozyme. Nature 414 (6859): 82–84. PMID 11689947. DOI: 10.1038/35102081.
- ↑ Sumper M, Luce R (January 1975). Evidence for de novo production of self-replicating and environmentally adapted RNA structures by bacteriophage Qbeta replicase. Proceedings of the National Academy of Sciences of the United States of America 72 (1): 162–166. PMID 1054493. PMC 432262. DOI: 10.1073/pnas.72.1.162.
- ↑ Mills DR, Peterson RL, Spiegelman S (July 1967). An extracellular Darwinian experiment with a self-duplicating nucleic acid molecule. Proceedings of the National Academy of Sciences of the United States of America 58 (1): 217–224. PMID 5231602. PMC 335620. DOI: 10.1073/pnas.58.1.217.
- ↑ Shaposhnikov GK (1966). Origin and breakdown of reproductive isolation and the criterion of the species.. Entomological Review 45: 1–8. Gearchiveerd van origineel op 8 september 2013.
- ↑ Rose MR (May 1984). Artificial Selection on a Fitness-Component in Drosophila Melanogaster. Evolution; International Journal of Organic Evolution 38 (3): 516–526. PMID 28555975. DOI: 10.2307/2408701.
- ↑ Rose MR, Passananti HB, Matos M (2004), Methuselah Flies. World Scientific, Singapore. DOI:10.1142/5457. ISBN 978-981-238-741-7.
- ↑ Burke MK, Dunham JP, Shahrestani P, Thornton KR, Rose MR, Long AD (September 2010). Genome-wide analysis of a long-term evolution experiment with Drosophila. Nature 467 (7315): 587–590. PMID 20844486. DOI: 10.1038/nature09352.
- ↑ Schlötterer C, Tobler R, Kofler R, Nolte V (November 2014). Sequencing pools of individuals - mining genome-wide polymorphism data without big funding. Nature Reviews. Genetics 15 (11): 749–763. PMID 25246196. DOI: 10.1038/nrg3803.
- ↑ Schlötterer C, Kofler R, Versace E, Tobler R, Franssen SU (May 2015). Combining experimental evolution with next-generation sequencing: a powerful tool to study adaptation from standing genetic variation. Heredity 114 (5): 431–440. PMID 25269380. PMC 4815507. DOI: 10.1038/hdy.2014.86.
- ↑ Zhou D, Xue J, Chen J, Morcillo P, Lambert JD, White KP, Haddad GG (May 2007). Experimental selection for Drosophila survival in extremely low O(2) environment. PLOS ONE 2 (5): e490. PMID 17534440. PMC 1871610. DOI: 10.1371/journal.pone.0000490.
- ↑ Zhou D, Udpa N, Gersten M, Visk DW, Bashir A, Xue J, Frazer KA, Posakony JW, Subramaniam S, Bafna V, Haddad GG (February 2011). Experimental selection of hypoxia-tolerant Drosophila melanogaster. Proceedings of the National Academy of Sciences of the United States of America 108 (6): 2349–2354. PMID 21262834. PMC 3038716. DOI: 10.1073/pnas.1010643108.
- ↑ Remolina SC, Chang PL, Leips J, Nuzhdin SV, Hughes KA (November 2012). Genomic basis of aging and life-history evolution in Drosophila melanogaster. Evolution; International Journal of Organic Evolution 66 (11): 3390–3403. PMID 23106705. PMC 4539122. DOI: 10.1111/j.1558-5646.2012.01710.x.
- ↑ Rainey PB, Travisano M (July 1998). Adaptive radiation in a heterogeneous environment. Nature 394 (6688): 69–72. PMID 9665128. DOI: 10.1038/27900.
- ↑ Chua SL, Ding Y, Liu Y, Cai Z, Zhou J, Swarup S, Drautz-Moses DI, Schuster SC, Kjelleberg S, Givskov M, Yang L (November 2016). Reactive oxygen species drive evolution of pro-biofilm variants in pathogens by modulating cyclic-di-GMP levels. Open Biology 6 (11): 160162. PMID 27881736. PMC 5133437. DOI: 10.1098/rsob.160162.
- ↑ (en) Ma Y, Chua SL (15 november 2021). Geen collaterale gevoeligheid voor antibiotica door het afwisselen van antibioticaparen. The Lancet Microbe 3 (1): e7. ISSN: 2666-5247. PMID 35544116. DOI: 10.1016/S2666-5247(21)00270-6.
- ↑ Rainey PB, Travisano M (July 1998). Adaptive radiation in a heterogeneous environment. Nature 394 (6688): 69–72. PMID 9665128. PMC 3758440. DOI: 10.1038/nature12344.
- ↑ Lenski RE, Rose MR, Simpson SC, Tadler SC (1 december 1991). Long-Term Experimental Evolution in Escherichia coli. I. Adaptation and Divergence During 2,000 Generations. The American Naturalist 138 (6): 1315–1341. ISSN: 0003-0147. DOI: 10.1086/285289.
- ↑ Fox JW, Lenski RE (June 2015). From Here to Eternity--The Theory and Practice of a Really Long Experiment. PLOS Biology 13 (6): e1002185. PMID 26102073. PMC 4477892. DOI: 10.1371/journal.pbio.1002185.
- ↑ Blount ZD, Borland CZ, Lenski RE (June 2008). Historical contingency and the evolution of a key innovation in an experimental population of Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America 105 (23): 7899–7906. PMID 18524956. PMC 2430337. DOI: 10.1073/pnas.0803151105.
- ↑ Lenski RE, E. coli Long-term Experimental Evolution Project Site. Michigan State University. Gearchiveerd op 27 juli 2017. Geraadpleegd op 8 juli 2004.
- ↑ (en) Bussotti, Giovanni, Piel, Laura, Pescher, Pascale, Domagalska, Malgorzata A., Rajan, K. Shanmugha (21 december 2021). Genome instability drives epistatic adaptation in the human pathogen Leishmania. Proceedings of the National Academy of Sciences 118 (51): e2113744118. ISSN: 0027-8424. PMID 34903666. PMC 8713814. DOI: 10.1073/pnas.2113744118.
- ↑ Swallow JG, Carter PA, Garland T (May 1998). Artificial selection for increased wheel-running behavior in house mice. Behavior Genetics 28 (3): 227–237. PMID 9670598. DOI: 10.1023/a:1021479331779. Gearchiveerd van origineel op 3 maart 2016.
- ↑ Keeney BK, Raichlen DA, Meek TH, Wijeratne RS, Middleton KM, Gerdeman GL, Garland T (December 2008). Differential response to a selective cannabinoid receptor antagonist (SR141716: rimonabant) in female mice from lines selectively bred for high voluntary wheel-running behaviour. Behavioural Pharmacology 19 (8): 812–820. PMID 19020416. DOI: 10.1097/FBP.0b013e32831c3b6b.
- ↑ Sadowska ET, Baliga-Klimczyk K, Chrzaścik KM, Koteja P (2008). Laboratory model of adaptive radiation: a selection experiment in the bank vole. Physiological and Biochemical Zoology 81 (5): 627–640. PMID 18781839. DOI: 10.1086/590164.
- ↑ Sadowska ET, Stawski C, Rudolf A, Dheyongera G, Chrząścik KM, Baliga-Klimczyk K, Koteja P (May 2015). Evolution of basal metabolic rate in bank voles from a multidirectional selection experiment. Proceedings. Biological Sciences 282 (1806): 20150025. PMID 25876844. PMC 4426621. DOI: 10.1098/rspb.2015.0025.
- ↑ Dheyongera G, Grzebyk K, Rudolf AM, Sadowska ET, Koteja P (April 2016). The effect of chlorpyrifos on thermogenic capacity of bank voles selected for increased aerobic exercise metabolism. Chemosphere 149: 383–390. PMID 26878110. DOI: 10.1016/j.chemosphere.2015.12.120.
- ↑ Maiti U, Sadowska ET, ChrzĄścik KM, Koteja P (August 2019). Experimental evolution of personality traits: open-field exploration in bank voles from a multidirectional selection experiment. Current Zoology 65 (4): 375–384. PMID 31413710. PMC 6688576. DOI: 10.1093/cz/zoy068.
- ↑ Kohl KD, Sadowska ET, Rudolf AM, Dearing MD, Koteja P (2016). Experimental Evolution on a Wild Mammal Species Results in Modifications of Gut Microbial Communities. Frontiers in Microbiology 7: 634. PMID 27199960. PMC 4854874. DOI: 10.3389/fmicb.2016.00634.
- ↑ Sleight SC, Bartley BA, Lieviant JA, Sauro HM (November 2010). Designing and engineering evolutionary robust genetic circuits. Journal of Biological Engineering 4: 12. PMID 21040586. PMC 2991278. DOI: 10.1186/1754-1611-4-12.
- ↑ González C, Ray JC, Manhart M, Adams RM, Nevozhay D, Morozov AV, Balázsi G (August 2015). Stress-response balance drives the evolution of a network module and its host genome. Molecular Systems Biology 11 (8): 827. PMID 26324468. PMC 4562500. DOI: 10.15252/msb.20156185.
- ↑ Kheir Gouda M, Manhart M, Balázsi G (December 2019). Evolutionary regain of lost gene circuit function. Proceedings of the National Academy of Sciences 116 (50): 25162–25171. PMID 31754027. PMC 6911209. DOI: 10.1073/pnas.1912257116.
- ↑ Farquhar KS, Charlebois DA, Szenk M, Cohen J, Nevozhay D, Balázsi G (June 2019). Role of network-mediated stochasticity in mammalian drug resistance. Nature Communications 10 (1): 2766. PMID 31235692. PMC 6591227. DOI: 10.1038/s41467-019-10330-w.
- ↑ Barrett RD, Paccard A, Healy TM, Bergek S, Schulte PM, Schluter D, Rogers SM (January 2011). Rapid evolution of cold tolerance in stickleback. Proceedings. Biological Sciences 278 (1703): 233–238. PMID 20685715. PMC 3013383. DOI: 10.1098/rspb.2010.0923.
- ↑ Dragosits M, Mattanovich D (July 2013). Adaptive laboratory evolution -- principles and applications for biotechnology. Microbial Cell Factories 12 (1): 64. PMID 23815749. PMC 3716822. DOI: 10.1186/1475-2859-12-64.
